Sono state determinate molte strutture tridimensionali delle proteine e sono stati desunti molti principi generali.
 La struttura di una proteina ha vari livelli. Struttura primaria, struttura secondaria e super-secondaria, struttura terziaria, struttura quaternaria.
La struttura di una proteina ha vari livelli. Struttura primaria, struttura secondaria e super-secondaria, struttura terziaria, struttura quaternaria.
Per Struttura primaria intendiamo la sequenza di amminoacidi nelle sue catene peptidiche, quale amminoacido viene prima e quale segue, ed ogni proteina ha una sequenza unica.
Qui di seguito è illustrata la sequenza degli amminoacidi dell’insulina umana che sono tenuti insieme da legami di tipo covalente chiamato legame ammidico o legame peptidico
A seconda della sequenza specifica di amminoacidi avremo alcuni tipi di strutture secondarie e possono essere presenti legami a disolfuro se si trovano a distanza opportune due cisteine
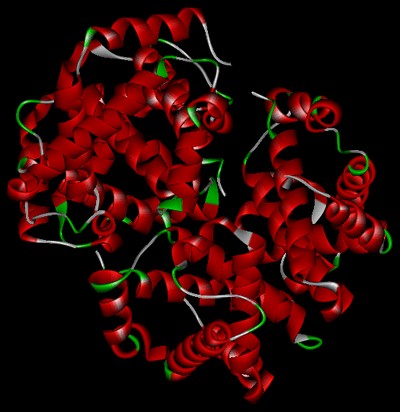
Struttura secondaria è l’arrangiamento spaziale che una sequenza di vari amminoacidi assume localmente. Le strutture ritrovate in natura sono l’alfa elica destrosa, la beta pieghe e la random coil, che sono tutte determinate dai particolari amminoacidi e dagli angoli psi e phi attorno al legame peptidico.
Esiste un altro livello locale chiamato Giro (Turn) che è una struttura secondaria organizzata ma non ripetitiva causata da legami ad H che non si propagano, come invece accade nella alfa elica.
L’organizzazione sempre locale di più strutture secondarie è chiamata Supersecondaria che ha sempre un compito e una funzione biologica. Questa si forma per la spontanea formazione di legami tra le strutture secondarie. Il tipo di legami qui coinvolti sono legami ad Idrogeno, legami di Van Der Walls e effetti idrofobici.
Struttura terziaria è la forma che l’intero peptide assume nello spazio con la formazione di legami ad H, ionici con il solvente acquoso, e con la formazione di legami di Van Der Walls ed effetti idrofobici all’interno della struttura terziaria.
Struttura quaternaria è la struttura nello spazio di proteine composte di due o più catene polipeptidiche denominate subunità. Questa struttura spiega la proprietà dell’allosteria, la modulazione della cinetica degli enzimi ed altre proprietà.
La proteina nella figura è formata da quattro subunità di tipo alfa e beta, associate con un gruppo prostetico Eme. E’ l’emoglobina
Strutture tridimensionali delle Proteine in Jmol
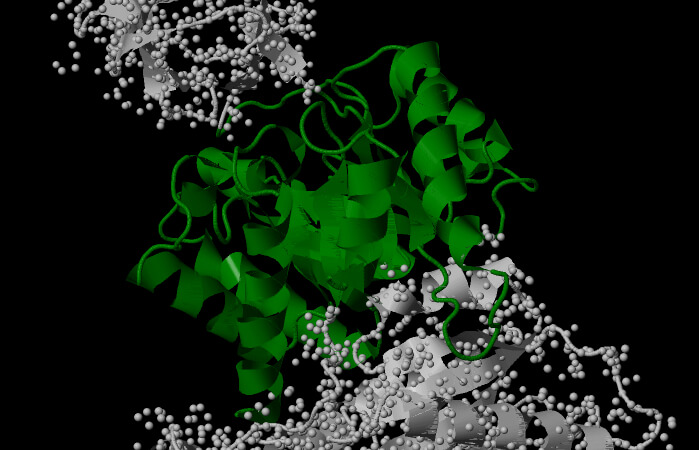
Qui visualizziamo le strutture tridimensionali secondarie, terziarie e quaternarie. In un’altra pagina entriamo nei dettagli delle singole strutture secondarie.
In quest’altra proteina, la ribonucleasi bovina A, troviamo le altre strutture secondarie..
– Qui sono mostrati tutti i legami ad H nelle tre strutture secondarie. Se si punta il mouse alle estremità dei segni dei legami ad H si possono individuare gli amminoacidi coinvolti e la loro posizione relativa nella catena.
Alfa elica Trattazione delle ALFA ELICHE – TRattazione delle Beta a Pieghe BETA A PIEGHE – Gomiti (random coil)
Una proteina che è possibile analizzare è la PAPAINA