Il processo della replicazione del DNA varia in modo notevole per complessità tra cellule procariote ed eucariote, ma ha in comune il fatto che il doppio filamento viene separato ed ogni singolo filamento viene replicato in modo complementare per creare una copia del tutto uguale al filamento originale.

Il filamento complementare generato è creato nella direzione che va da 5’ al 3’ in quanto la DNA Polimerasi può aggiungere un nucleotide solo sull’OH 3’ libero del filamento.
Poiché i due filamenti sono complementari e corrono in direzione opposta (5’→3’ e 3’→ 5’), avranno due modalità diverse di replicazione. Un filamento fa da Stampo e l’altro viene definito Ritardato
Alcune DNA Polimerasi hanno la possibilità di fare una correzione di bozza sul filamento generato ed usare una esonucleasi per rimuovere e rimpiazzare nucleotidi aggiunti per errore.
Questo fatto rende le DNA polimerasi uno degli enzimi più accurati poiché fa solo un errore su 1 milione di basi aggiunte.
Queste polimerasi sono divise in 7 famiglie secondo l’omologia delle sequenze e le similarità nella struttura tridimensionale.
Perché le DNA polimerasi possano esplicare la loro azione, è necessario che altri enzimi e proteine agiscano per liberare e distaccare i due filamenti da duplicare. La doppia elica deve essere prima svolta, poi devono essere separati i due filamenti e tenuti in questa posizione, poi successivamente la DNA Primasi deve porre un RNA primer sul filamento template e dare il segnale della replicazione.
La replicazione si basa sull’aggiunta di un nucleotide all’estremità 3’ che sia complementare al nucleoside sul filamento template secondo le accoppiate delle basi azotate C-G e A-T, Citosina Guanina, Adenina Timina
La reazione che si può descrivere così produce uno ione difosfato ed un nuovo legame fosfodiestere
(DNA)n residui + dNTP → (DNA)n+1 residui + PPi
La struttura fondamentale di tutte le DNA Polimerasi è di avere sottodomini chiamati Palmo, Dita e Pollice ed assomigliano ad una mano destra aperta.
Il Palmo contiene gli amminoacidi che fatto la catalisi nel sito attivo. Le Dita sono importanti nel riconoscimento del nucleotide e nel legame. Il Pollice è importante nel legare il substrato DNA.
Tra il dito e il pollice c’è una sacca che è fatta di due regioni; il sito d’inserzione e quello di post inserzione.
I nucleotidi che entrano si legano al sito di inserzione e la nuova coppia di basi risiede nel sito postinserzione.
Gran parte delle DNA Polimerasi vanno con un meccanismo a due ioni metallici in cui due ioni nel sito attivo lavorano per stabilizzare lo stato di transizione pentacoordinato e permettere l’attacco al P della base entrante.
Le DNA Polimerasi sono anche definite come trascrittasi, oppure DNA Polimerasi DNA Dipendenti. La Trascrittasi inversa è invece una DNA Polimerasi RNA dipendente perché sintetizza il DNA a partire da un filamento di RNA
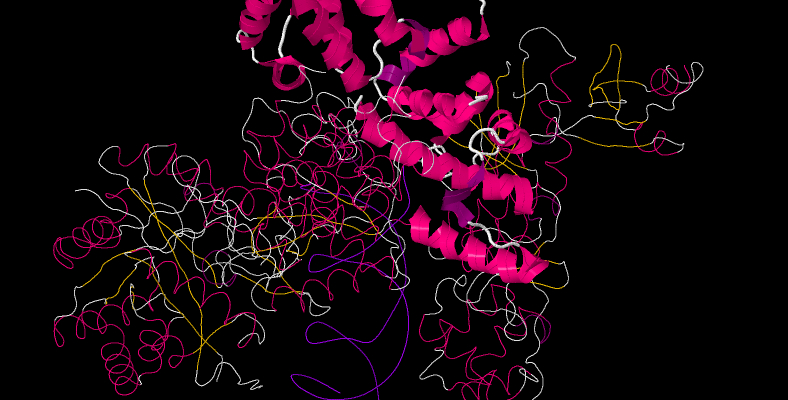
Di seguito viene descritta la DNA Polimerasi III del Thermus Aquaticus che si può leggere in originale a Proteopedia
A questo oloenzima è legato un DNA primer template ed un desossinucleotide entrante. L’interazione con DNA avviene nel solco minore del DNA attraverso le interazioni dei domini delle dita, pollice e dominio beta.
Il dominio delle dita, che fa parte del sito attivo, seleziona in base allo zucchero presente il desossiribonucleotide rispetto al ribonucleotide (del RNA) posizionandolo nella orientazione corretta per la catalisi.
Il dominio del Palmo contiene il sito catalitico per l’addizione del nucleotide compreso tre aspartati ben conservati (463,465,618) Esso aiuta al posizionamento del gruppo OH 3′ del primer e i deossinucleotidi entranti in modo che la base coniugata sia nella orientazione corretta per la catalisi.
Il dominio del Pollice forma una parte del canale di uscita del DNA e contiene molti residui che interagiscono con esso.
La lega il clamp beta ed aiuta a posizionare il DNA a doppia elica.
La contiene una piega di legame di oligonucleotide che avvolge il DNA a singolo filamento in una scanalatura di superficie.
Questo è il desossinucleotide che entra che deve sottostare alla selezione in base allo zucchero dalla Hys 815 e Tyr 821 in verde, all’orientamento e posizionamento, stabilizzazione, catalisi ed addizione alla catena nascente di DNA.
Il , della Polimerasi e istidinol fosfatasi, forma una parte del canale di uscita del DNA.