La ricerca di medicinali antivirali ha portato ad esplorare nuovi obiettivi di attacco al SARS-CoV2 differenti dalla proteina SPIKE quali la proteasi cisteinica virale MPro.
Questa proteina cisteina virale MPro è codificata dal genoma di 30mila nucleotidi del Coronavirus ed è essenziale nella replicazione e trascrizione del virus stesso.
Dal primo processo di traduzione del genoma virale sono prodotti due polipeptidi che per proteolisi originano poi tra l’altro la proteasi cisteinica virale Mpro, da 33.8 kDalton, che è simile alla chimotripsina, ma non ha omologhi tra le proteasi umane.
Proprio questa unicità della proteasi cisteinica virale Mpro la rende obiettivo di ricerche di nuovi medicinali ed inibitori specifici, anche perché si è visto che la MPro è altamente conservata nell’evoluzione.
La Mpro per prima si separa dai due polipeptidi per autolisi e poi con 11 siti specifici di attacco produce le altre proteine utili alla replicazione e traduzione del Coronavirus stesso.
La diade catalitica della proteasi cisteina virale è composta dalla CIsteina145 e Istidina41 che si trovano nel sito attivo costituito da 4 sacche principali.
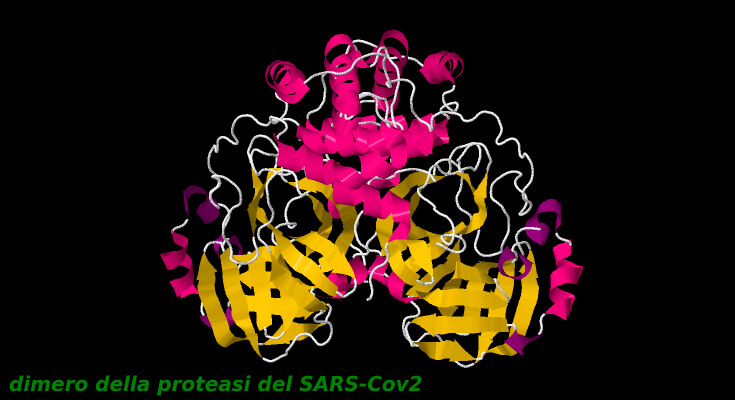
Il sito attivo si trova in una sacca tra due domini N terminali della struttura a tre domini del Monomero, mentre il dominio della elica C terminale è coinvolto nella regolazione e dimerizzazione dell’enzima.
L’enzima infatti è un omodimero, costituito da due unità monomeriche identiche da 306 amminoacidi che da sole non hanno attività catalitica.
“In generale l’attività enzimatica della Mpro è legata all’architettura del sito attivo, che dipende in modo critico dalla dimerizzazione dell’enzima e dal corretto orientamento relativo dei sottodomini. Questo potrebbe permettere a dei leganti che si legano all’esterno del sito attivo di modificare la sua attività. Abbiamo infatti identificato due siti allosterici del Mpro” (science.org)
Sono stati identificati cinque molecole che si legano ad una sacca idrofobica nel dominio di dimerizzazione, posizionati vicini alla buca dell’ossianione nella sacca S1 del sito di legame. Uno di questi composti mostrava forte attività antivirale. Sacca idrofobica formata da Ile213, Leu253, Gln256, Val297, e Cys300. (vedere sotto l’immagine in 3d)
Ogni monomero è formato da tre domini che sono: dominio I dall’AA 1 al 101 ed in VERDE nell’immagine in 3d, mentre dominio II dal 102 al 184 in ROSA e il dominio III dal 201 al 306 in ROSSO. In Blu c’è il dominio N terminale al cui inizio è posta una Serina.
I domini I e II sono formati da una struttura a barile Beta con legami ad H in antiparallelo, mentre il dominio III è formato da eliche destrose.
Di seguito sono mostrate le strutture delle nuove molecole sperimentate come inibitrici della Mpro e possibili farmaci antivirali
– –
–
– – – molecola anticancro usata per il cancro del colon retto. Forma un tioestere con la Cisteina del sito catalitico.